摘要

关键词
- 基因编辑 (Base Editing)
- T细胞功能 (T Cell Functions)
- 突变体图谱 (Mutagenesis Map)
- 免疫疗法 (Immunotherapy)
- 高通量筛选 (High-Throughput Screening)
- 分子功能调控 (Molecular Functional Tuning)
研究背景
T细胞在免疫应答中起关键作用,其功能的异常可导致自免疫疾病、炎症或免疫缺陷。虽然CRISPR筛选已经发现了一些控制T细胞功能的基因,但对关键基因中特定核苷酸的功能研究仍不够深入。本研究利用基因编辑技术,在不引起双链断裂的情况下实现高效的目标突变,通过大规模筛选揭示了调控T细胞激活和功能的分子机制,并验证了这些突变对T细胞细胞毒性和激活功能的调控能力。
创新点
- 提出了一种高通量基因编辑筛选平台,揭示T细胞功能调控的关键突变位点。
- 通过基因编辑技术精确定位蛋白结构域及其相互作用位点。
- 验证了突变对T细胞激活和细胞因子分泌的双向调控能力。
研究内容
本研究开发了一个优化的腺嘌呤和胞嘧啶基因编辑平台,通过慢病毒系统将编辑工具导入人类T细胞,靶向385个与T细胞功能相关的基因编码区,构建了约117,000个导向RNA库。通过流式细胞术筛选,研究发现了一系列蛋白结构域和氨基酸残基,这些残基的突变能够显著影响T细胞的激活状态和细胞因子分泌。
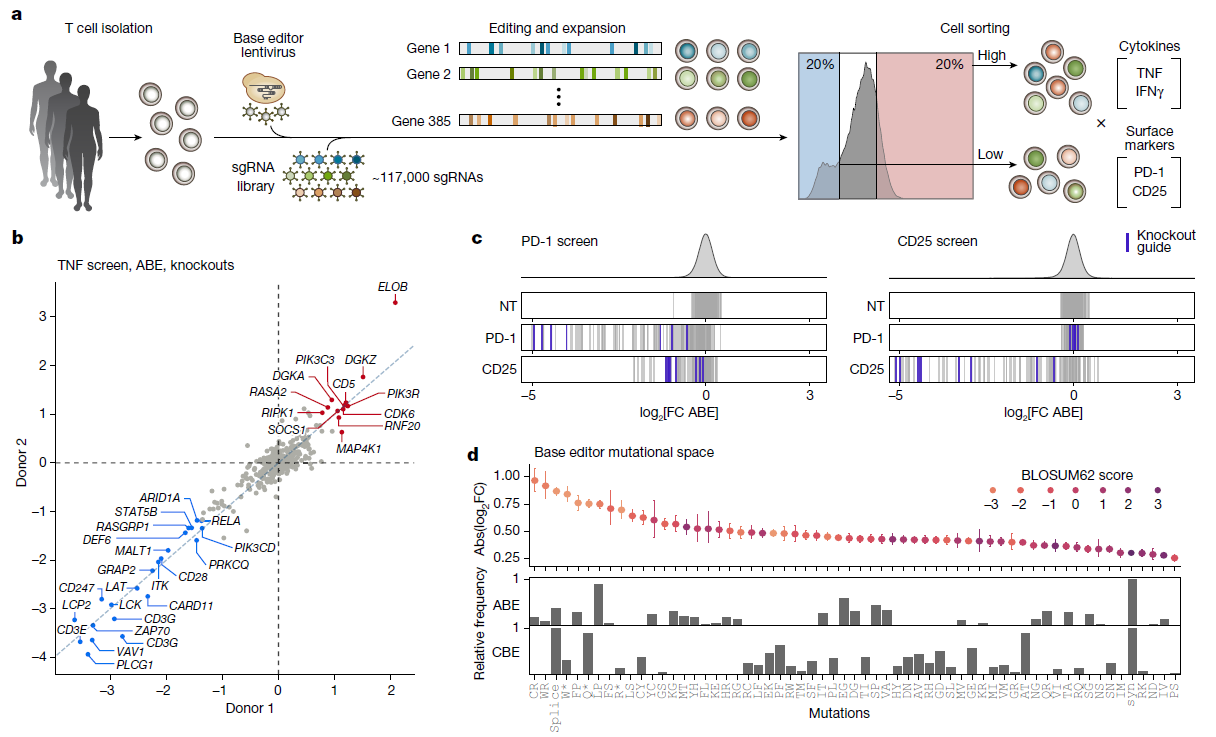
(a) 在三个供体的原代T细胞中进行ABE和CBE基础编辑筛选的示意图。
(b) 散点图展示了平均log2转化的折叠变化(log2FC),对每个基因而言,比较了在两个供体中由前三个诱导敲除的sgRNAs(破坏起始密码子或编辑剪接位点)造成的平均效果。负调节因子以红色显示,正调节因子以蓝色显示。
(c) 针对PD-1或CD25的细胞表面表达筛选的log2FC值(高/低排序分组),展示了直接靶向PD-1或CD25的单导向RNA(竖线)或非靶向对照的变化。竖线表示预计导致功能性敲除的导向。
(d) 顶部:ABE和CBE筛选中展示的TNF生成相关基因中不同突变类型的效果范围,按生物替换可能性(BLOSUM62得分)着色。中部和底部分别显示ABE和CBE筛选中每种突变类型的相对频率。
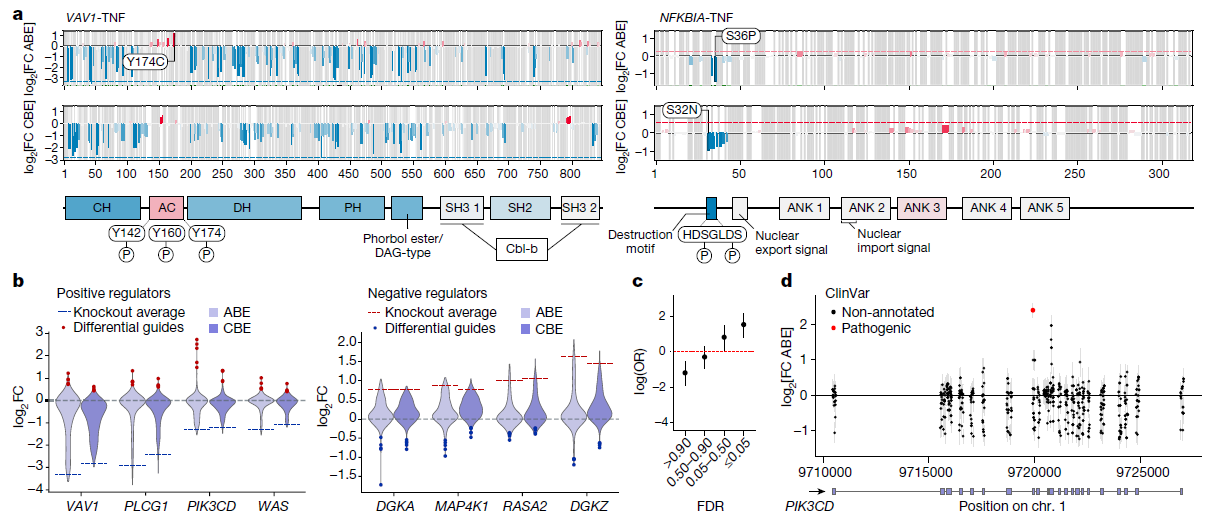
(a) 非终止的非同义基础编辑对TNF生产的平均log2FC效应图,覆盖了VAV1(左)和NFKBIA(右)的开放阅读框。
(b) 小提琴图展示了对TNF的正调节基因和负调节基因的ABE和CBE导向的log2FC分布。
(c) 在TNF ABE筛选中,基础编辑变体根据MAGeCK导向水平的假发现率(FDR)分组,绘制了与ClinVar中“致病”变体重叠的比值对数比。
(d) 基础编辑变体的结果展示了在PIK3CD位点的突变,其中与ClinVar中“致病”位点匹配的用红色显示。
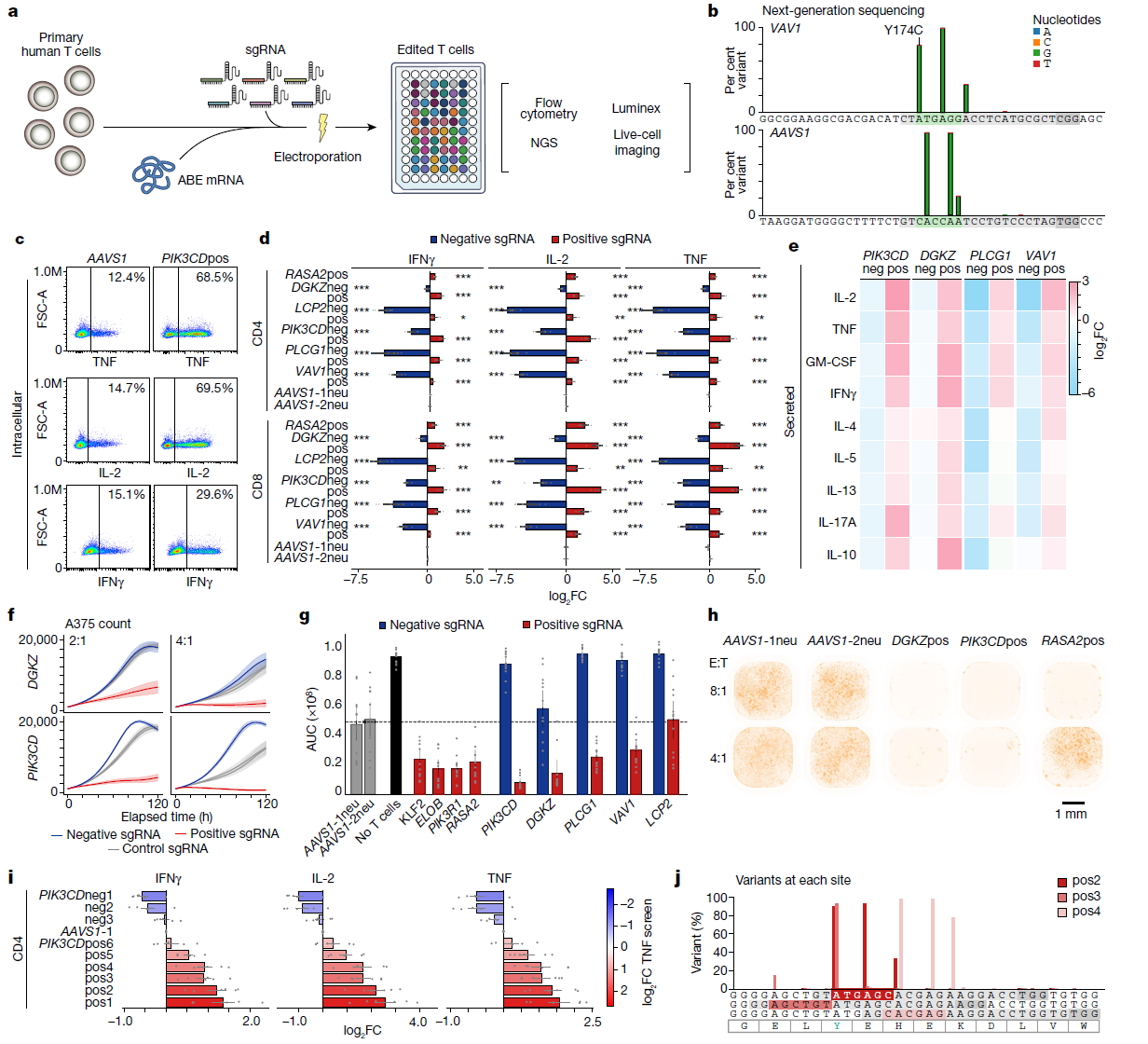
(a) 示意图展示了通过共电穿孔实现ABE mRNA和合成sgRNA的单次实验验证流程。
(b) 在VAV1的阳性位点(VAV1pos sgRNA)和AAVS1对照位点的基础编辑,由深度扩增子测序验证,并通过Crispresso2分析。绿色区域表示预测的编辑窗口,灰色表示导向序列,深灰色表示NGG PAM。
(c) 代表性流式细胞仪图,展示了在对照(AAVS1)或PIK3CDpos sgRNA编辑的T细胞中,指定细胞因子的表达情况。分析针对CD4+ T细胞群体。
(d) 测量的细胞内指定细胞因子表达的log2倍数变化(相对于对照,两种AAVS1导向RNA的均值),通过流式细胞术获得。原始筛选中的负性sgRNA以蓝色表示,阳性sgRNA以红色表示。n = 6;*P < 0.05,**P < 0.01,***P < 0.001。阳性(pos)和负性(neg)分别表示对T细胞激活反应具有正效应和负效应的sgRNA。
(e) 用Luminex测量基础编辑T细胞培养上清中的细胞因子分泌情况。热图表示相对于两种AAVS1对照的细胞log2倍数变化。n = 4。
(f) 基础编辑的特异性T细胞对A375细胞的细胞杀伤能力,通过使用靶向DGKZ或PIK3CD的阳性、负性导向RNA或对照RNA测量,并通过Incucyte成像随时间记录。n = 6。
(g) 在效应细胞与靶细胞比例(E:T)为4时,与基础编辑T细胞共同培养的A375细胞计数随时间变化的曲线下面积(AUC)。n = 6名供体,每个供体技术重复两次。
(h) 使用指示性导向RNA和E:T比率在120小时后共培养的A375细胞与基础编辑的抗原特异性T细胞的成像结果。
(i) 在CD4+ T细胞中基于阵列验证的PIK3CD九个导向RNA的指定细胞因子生产变化,相对于AAVS1对照;颜色表示原始ABE TNF筛选中的log2倍数变化值。n = 6。
(j) 聚集在Y524附近的基础编辑突变(绿色)通过测序显示出不同的突变形式。所有n值均表示人类供体数量。数据以均值±标准误表示。双尾独立双样本t检验。
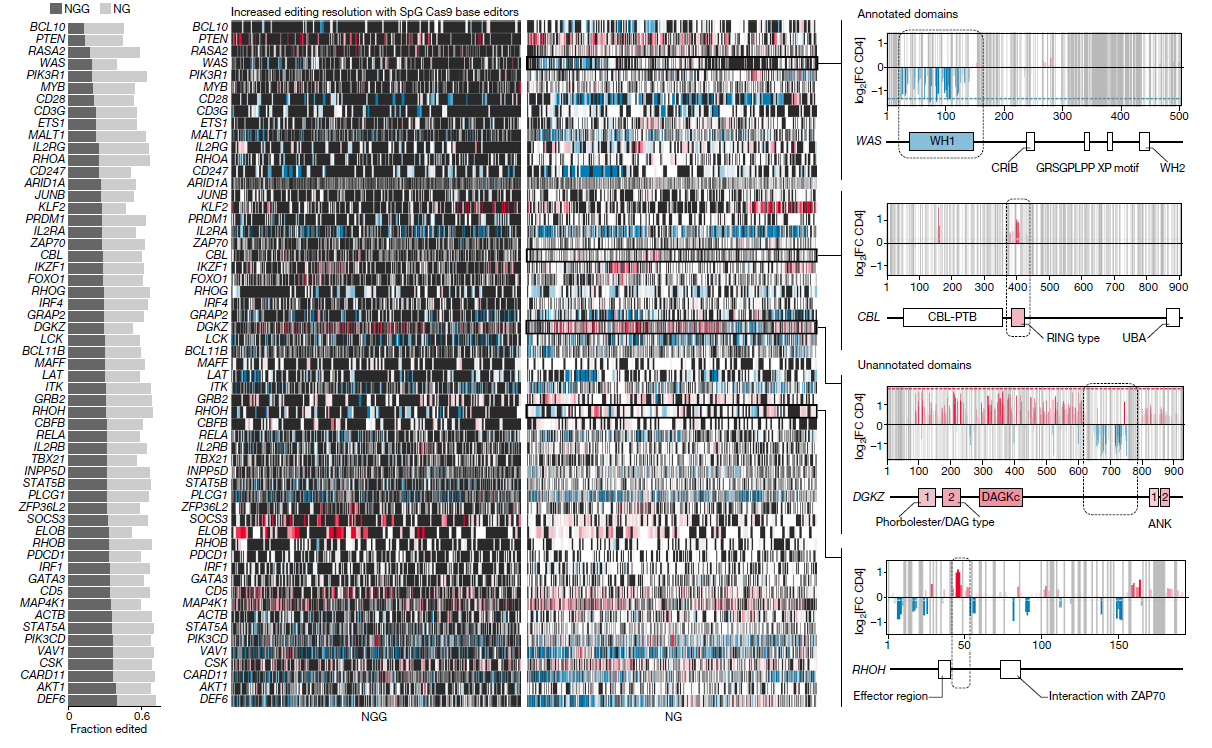
(a) 比较了基于NGG和NG PAM的基础编辑分辨率,展示了对57个基因的影响范围。
(b) 基础编辑在不同基因(例如WAS、CBL、DGKZ和RHOH)中揭示的功能区域。
(c) 热图详细显示了每个基因中编辑效果,按功能域划分的颜色展示突变的正负效应。
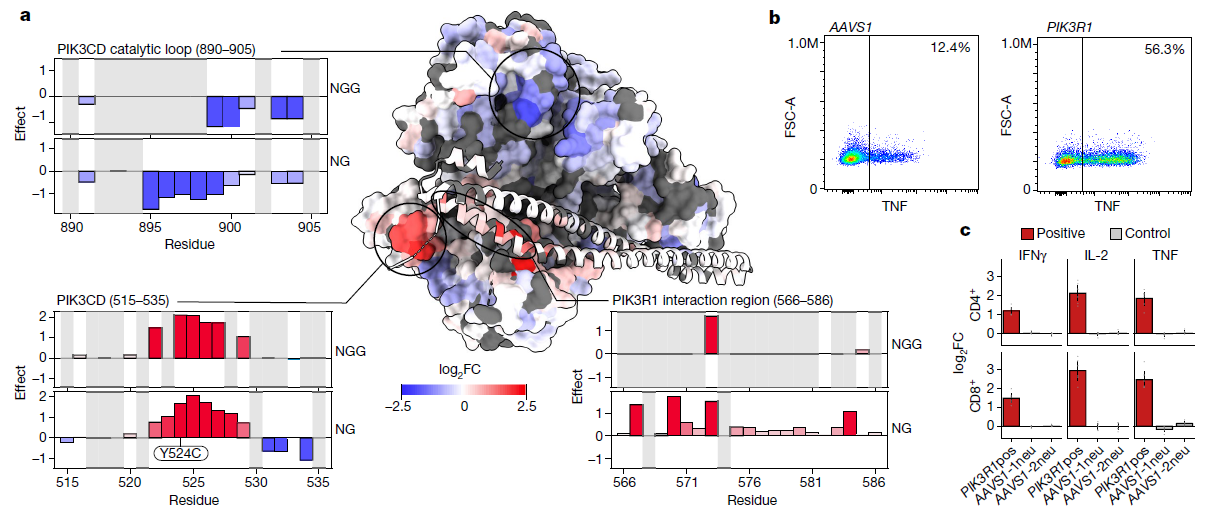
(a) PIK3CD-PIK3R1复合物的结构模型,基于基础编辑的log2FC效应对残基着色。
(b) 流式细胞仪图展示了用PIK3R1或对照导向编辑的T细胞中TNF的分布。
(c) CD4+或CD8+ T细胞中阳性PIK3R1导向对细胞因子生成的log2FC。
结论与展望
本研究展示了基于基因编辑的突变筛选技术在调控T细胞功能中的潜力,发现了一系列能够调控T细胞激活和免疫反应的功能性突变,为免疫疗法的设计提供了重要参考。未来研究可进一步拓展至非编码序列及其他免疫细胞类型,促进疾病相关基因变异的解析。
论文直达
原文标题:Base-editing mutagenesis maps alleles to tune human T cell functions
Nature, 2024, 625, 805–812.
点击以下链接阅读原文:

